呼吸道感染常见于成人和儿童,具有发病率高、致死率高的特点,严重威胁着公共卫生安全。其病原组成复杂、变异频繁,往往还存在复杂的共感染,因此,进行及时准确的病原学诊断对临床上的诊断治疗至关重要【1】。临床病原学检测有多种方法,近年来分子生物学检测技术发展迅速,包括普通聚合酶链反应(PCR)、多重PCR、实时定量PCR和二代基因测序技术(NGS)等。NGS的发展为病原体的诊断带来了新的突破,临床上应用最多的是宏基因组二代测序技术(mNGS)。mNGS具有测序深度大、覆盖面广、测序效率高的有点,同时也存在着一定的局限性,如检测费用高、检测结果易受样本中人源序列干扰、DNA和RNA病原体不能同时检测、结果解读困难等【2】。如今,靶向高通量测序(tNGS)技术实现了病原富集技术和高通量测序技术的结合,具有正向富集、兼顾广谱性和高灵敏度等优势,受到广泛认可与应用。
tNGS是一种针对特定基因序列的靶向高通量测序技术,通过对目标基因序列设计特异性引物或探针进行超多重PCR扩增或探针捕获,然后进行高通量测序并进行生信分析,从而实现对样本中核酸的高灵敏及高分辨率的识别。核酸经此富集过程,人源序列的干扰显著降低,同时测序深度和覆盖度有充分的保证。tNGS主要分为两种方法:基于超多重PCR 扩增的扩增子测序和基于探针杂交的杂交捕获测序【3】。。扩增子测序的优势体现在起始量需求和检测下限,而杂交捕获测序的优势体现在高通量性和广谱性。研究发现,对高病毒载量样本,两种方法均有很好的富集效果;而对低病毒载量样本,多重PCR扩增子测序的富集效果明显优于探针捕获测序,可达10~1000倍【4】。
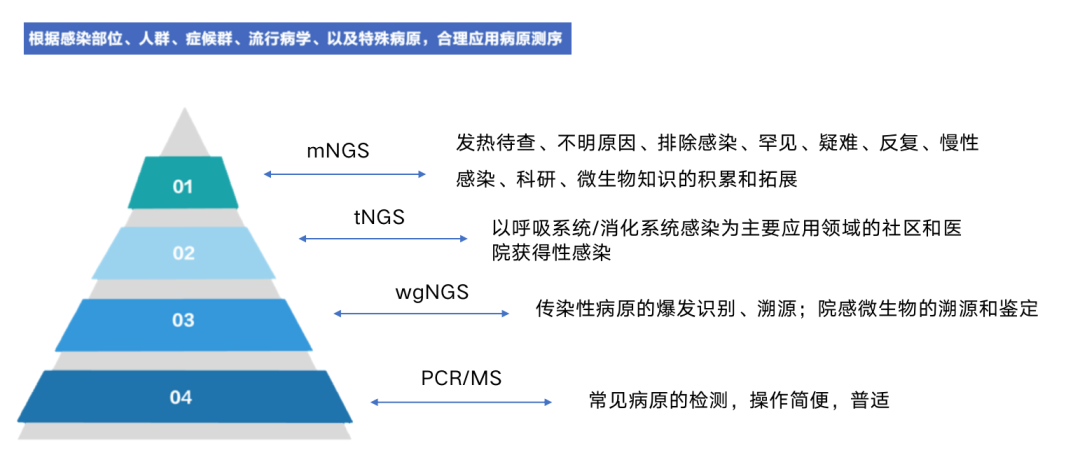
tNGS灵敏度较高,实现了DNA和RNA的共检,且无需预判病原体就能进行定量
tNGS技术靶向富集目标病原,提升检测灵敏度,还能排除宿主核酸的干扰,提升检测可信度【5】。一研究发现,与培养法相比,tNGS技术在呼吸道病原体检测中的灵敏度更高【6】。又一研究创建了一个靶向序列捕获面板ViroCap,对两组样本进行mNGS和tNGS的测序。一组来自提交给诊断病毒学实验室的样本,另一组来自儿童发烧研究中收集的样本。捕获后检出病毒的中位数倍数为674倍和296倍。第一组捕获后的覆盖度中位数从2.1%提升到83.2%,第二组从2.0%提升到75.6%。总之,在病原检测范围内,tNGS的灵敏度更高,检测结果的可信度也更高【7】。
《下呼吸道感染宏基因组二代测序报告临床解读路径专家共识》中提到:对于下呼吸道感染病原体的检测,tNGS的灵敏度和特异度都比mNGS高,所用检测时间更短,成本也更低【8】。一研究发现,tNGS和mNGS对下呼吸道感染标本的微生物检出率无显著差异(82.17% vs. 86.51%,P=0.41)。mNGS的检测谱更广、病原覆盖面更全,但tNGS能在一次检测中兼顾DNA/RNA型病原体,更具卫生经济学价值【9】。
病原靶向测序(tNGS)通过超多重PCR扩增与高通量测序两种技术的结合,能够对待测样本中几十种至几百种已知病原微生物及其毒力和/或耐药基因进行检测。对低浓度的病原微生物的检测,特别是其毒力和/或耐药基因检测,与病原宏基因组测序(mNGS)相比,tNGS具有病原谱范围明确、测序成本低等优势。因此,tNGS在临床检验领域正受到越来越多的关注(引自中国食品药品检定研究院)。一研究进行耐药基因检测,用tNGS发现在13种潜在病原体中,有7种(53.8%)的抗菌药物敏感性测试结果完全(或部分)一致【10】。另一研究发现,tNGS在22名颅内感染儿童中的18名中发现了致病性感染,阳性率为81.82%。在所有感染儿童中,革兰氏阴性菌检出率最高,其次是革兰氏阳性菌,而病毒的比例最低。该研究中tNGS的诊断效力高于传统测定,其AUC为0.794(传统测定AUC为 0.568),显著优于传统测定【11】。
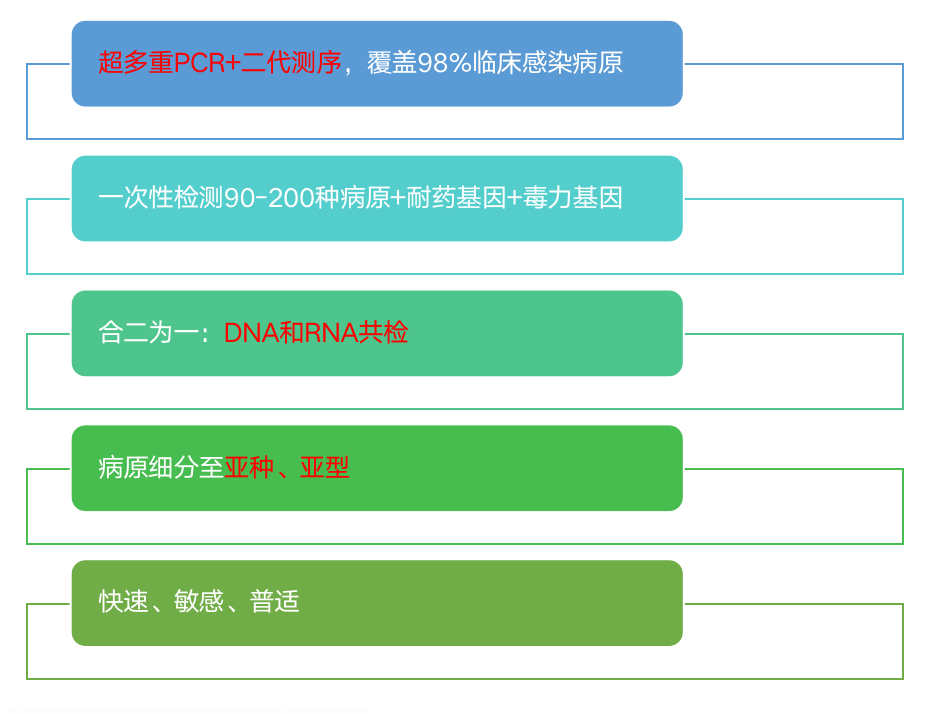
因此,采用靶向高通量测序 (tNGS) 技术,我们实现了有效性和性价比的平衡,提升精准防控感染的技术可及性,从而为更多呼吸道感染患者精准诊疗提供另一种选择。
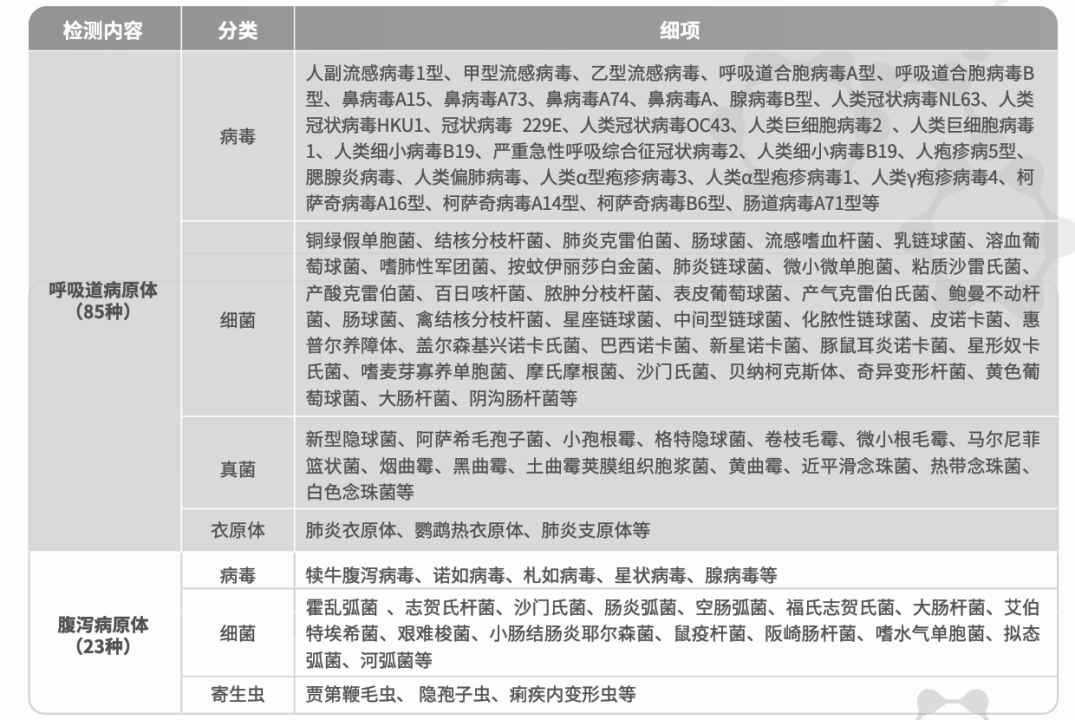
北京微未来tNGS高通量测序解决方案检测内容
-
采用超多重PCR (2000+) 建库体系,针对不同感染症候群目标病原及耐药基因设计特异性引物,对目标序列进行靶向扩增富集
-
利用核心算法及模型,应用二代测序技术,进行靶向高通量定量测序(Q-tNGS),实现病原广谱精准检测
DNA和RNA病原检测合二为一,病原细分至亚型、亚种
在病原微生物检测技术众多的如今,如何选择合适的检测方法,以期更好地解决临床问题,是实现感染性疾病精准诊疗的重要论题。
tNGS可实现根据临床需求设计检测范围,能对待测样本中几百种已知病原微生物及其毒力和耐药基因进行检测,尤其是对低浓度病原微生物的检测,与mNGS相比,具有病原谱范围明确、测序成本低等优势。此外,tNGS聚焦于核心病原并分级呈现,降低了报告解读难度。因此,tNGS有着广泛的应用前景,在推动感染性疾病精准诊疗方面发挥重要作用。
参考文献
【1】GBD 2019 Diseases and Injuries Collaborators. Global burden of 369 diseases and injuries in 204 countries and territories, 1990- 2019: a systematic analysis for the Global Burden of Disease Study 2019. Lancet, 2020, 396(10258): 1204-1222. Erratum in: Lancet, 2020, 396(10262): 1562.
【2】Miao Q, Ma Y, Wang Q, Pan J, Zhang Y, Jin W, Yao Y, Su Y, Huang Y, Wang M, Li B, Li H, Zhou C, Li C, Ye M, Xu X, Li Y, Hu B. Microbiological Diagnostic Performance of Metagenomic Next-generation Sequencing When Applied to Clinical Practice. Clin Infect Dis. 2018 Nov 13;67(suppl_2):S231-S240. doi: 10.1093/cid/ciy693. PMID: 30423048.
【3】Hilt EE, Ferrieri P. Next Generation and Other Sequencing Technologies in Diagnostic Microbiology and Infectious Diseases. Genes (Basel). 2022 Aug 31;13(9):1566. doi: 10.3390/genes13091566. PMID: 36140733; PMCID: PMC9498426.
【4】Xiao M, Liu X, Ji J, Li M, Li J, Yang L, Sun W, Ren P, Yang G, Zhao J, Liang T, Ren H, Chen T, Zhong H, Song W, Wang Y, Deng Z, Zhao Y, Ou Z, Wang D, Cai J, Cheng X, Feng T, Wu H, Gong Y, Yang H, Wang J, Xu X, Zhu S, Chen F, Zhang Y, Chen W, Li Y, Li J. Multiple approaches for massively parallel sequencing of SARS-CoV-2 genomes directly from clinical samples. Genome Med. 2020 Jun 30;12(1):57. doi: 10.1186/s13073-020-00751-4. PMID: 32605661; PMCID: PMC7325194.
【5】Guo Y, Li H, Chen H, Li Z, Ding W, Wang J, Yin Y, Jin L, Sun S, Jing C, Wang H. Metagenomic next-generation sequencing to identify pathogens and cancer in lung biopsy tissue. EBioMedicine. 2021 Nov;73:103639. doi: 10.1016/j.ebiom.2021.103639. Epub 2021 Oct 23. PMID: 34700283; PMCID: PMC8554462.
【6】Dai Y, Sheng K, Hu L. Diagnostic efficacy of targeted high-throughput sequencing for lower respiratory infection in preterm infants. Am J Transl Res. 2022 Nov 15;14(11):8204-8214. PMID: 36505277; PMCID: PMC9730095.
【7】Wylie TN, Wylie KM, Herter BN, Storch GA. Enhanced virome sequencing using targeted sequence capture. Genome Res. 2015 Dec;25(12):1910-20. doi: 10.1101/gr.191049.115. Epub 2015 Sep 22. PMID: 26395152; PMCID: PMC4665012.
【8】中华医学会呼吸病学分会. 下呼吸道感染宏基因组二代测序报告临床解读路径专家共识[J]. 中华结核和呼吸杂志,2023,46(04):322-335. DOI:10.3760/cma.j.cn112147-20220701-00553
【9】Li S, Tong J, Liu Y, Shen W, Hu P. Targeted next generation sequencing is comparable with metagenomic next generation sequencing in adults with pneumonia for pathogenic microorganism detection. J Infect. 2022 Nov;85(5):e127-e129. doi: 10.1016/j.jinf.2022.08.022. Epub 2022 Aug 26. PMID: 36031154.
【10】Gaston DC, Miller HB, Fissel JA, Jacobs E, Gough E, Wu J, Klein EY, Carroll KC, Simner PJ. Evaluation of Metagenomic and Targeted Next-Generation Sequencing Workflows for Detection of Respiratory Pathogens from Bronchoalveolar Lavage Fluid Specimens. J Clin Microbiol. 2022 Jul 20;60(7):e0052622. doi: 10.1128/jcm.00526-22. Epub 2022 Jun 13.PMID: 35695488; PMCID: PMC9297812.
【11】Li J, Zhang L, Yang X, Wang P, Feng L, Guo E, Chen Y. Diagnostic Significance of Targeted Next-Generation Sequencing in Central Nervous System Infections in Neurosurgery of Pediatrics. Infect Drug Resist. 2023 Apr 15;16:2227-2236. doi: 10.2147/IDR.S404277. PMID: 37090034; PMCID: PMC10115197.
本篇文章来源于微信公众号: 北京微未来



